Vantaggi: miglioramento di tutti i parametri della reazione.
• Lo stampo non è un plasmide o un fago con l'inserto di interesse ma un prodotto di PCR.
Bastano 30-90 ng di DNA
• Biochimica della
reazione: si applica il principio della PCR.
Si ha denaturazione dello
stampo e riutilizzo del primer (non si avrà crescita logaritmica
ma lineare)
Amplitaq DNA Polimerasi FS (Fluorescing Sequencing):
Taq modificata con
l'ingegneria genetica, con mutazione puntiforme nel sito attivo
per conferire buona affinità per i dideossinucleotidi,
e una mutazione puntiforme nel
dominio amino terminale che elimina l’attività nucleasica
5´-->3´.
Cycle sequencing: la reazione
avviene in una sola provetta usando 4 diversi fluorescenti come
marcatori.
Non marco il primer ma i
nucleotidi (per es. dTTP = fluorocromo rosso).
• Gel: Polimero (POP6)
Si versa il gel in un tubo capillare.
• Rilevazione: avviene
in modo automatico con il lettore ottico (laser) che legge i
nucleotidi fluorescenti.
Il laser rivela la diversa
fluorescenza (emissione a diversa lunghezza d’onda) per ogni
molecola stratificata nel capillare.
• Coloranti:
Big Dye (diclororodamine).
Contengono un sito donatore
fluorescinato legato ad uno dei 4 coloranti accettori
diclororodamine (dRhodamine).
La massima eccitazione di ogni
colorante è quella della fluorescina (donatore) e lo spettro di
emissione è quello della rodamina (accettore).
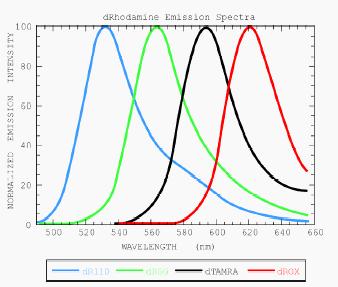
Hanno dei vantaggi rispetto alle rodamine usate in precedenza. Danno picchi più ristretti e più alti con riduzione delle sovrapposizioni degli spettri di emissione e quindi meno rumore di fondo nella lettura.

Spettro di emissione delle rhodamine:

Spettro di emissione delle dRhodamine (picchi più ravvicinati)
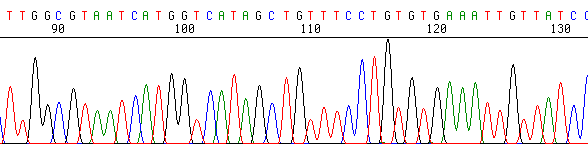
• Lettura: il software legge il segnale e lo trasforma in file di testo.
Elabora i dati, trasmessi dalla CCD camera, e per confronto con una matrice (già impostata) rimanda i risultati sotto forma di picchi.

4 colori/1 lane (seq. automatico) vs. 1 colore/4 lane.
Fonte
per i problemi: CRIBI
- Università di Padova